
人SNP芯片
产品名称: 人SNP芯片
英文名称: Human SNP Array
产品编号:
产品价格: 电话咨询:021-58955370
产品产地: null
品牌商标: null
更新时间: 2024-12-19T16:11:10
使用范围: null
- 联系人 : 付先生
- 地址 : 浦东新区张江高科技园区李冰路151号
- 邮编 : 201203
- 所在区域 : 上海
- 电话 : 177****9967 点击查看
- 传真 : 点击查看
- 邮箱 : market@shbio.com
- 二维码 : 点击查看
技术介绍
人类基因组由 30 亿个核苷酸组成,它们携带着人类的遗传信息并决定人体的生理特征。人类基因组序列的 0.1%—0.2% 在人种、人群和个体之间存在 DNA 序列的差异,即单核苷酸多态性(SNP,single nucleotide polymorphism)。许多的这些单核苷酸多态性可引起不同的遗传性状,即遗传的多态性(polymorphism),如 ABO 血型位点标记,白细胞 HLA 位点标记和个体药物代谢差异等。了解这些 DNA 序列的差异和单核苷酸多态性以及这些差异所表现的意义将给疾病的预测、诊断、预后和预防带来革命性的变化。
芯片推荐
1、Genome-Wide Human SNP Array 6.0
Affymetrix Genome-Wide Human SNP 6.0 芯片产品涵盖超过 1,800,000 个遗传变异标志物:包括超过 906,600 个 SNP 和超过 946,000 个用于检测拷贝数变化(CNV,Copy Number Variation)的探针:
▲482,000 个 SNP 来自于前代产品 500K 和 SNP5.0 芯片;
▲424,000 个 SNP 包括国际 HapMap 计划中的标签 SNP,X,Y 染色体和线粒体上具有代表性的 SNP, 以及来自于重组热点区域和 500K 芯片设计完成后新加入 dbSNP 数据库的 SNP;
▲202,000 个用于检测 5,677 个已知拷贝数变异区域的探针,这些区域来源于多基因组变异体数据库。该数据库中 3,182 个非重叠片断区域平均用 61 个探针来检测;
744,000 个探针平均分配到整个基因组上,用来发现未知的拷贝数变异区域。
2、Human Omni ZhongHua-8 BeadChip 芯片服务—特别覆盖中国人特有的常见和稀有变异
▲第一款人类种群特异的全基因组芯片,经过优化的标签 SNP 内容来自 HapMap 所有三个阶段以及千人基因组计划(1kGP),可用于在中国人种群中探索全新的疾病和性状关联。
▲特别覆盖中国人 81% 的常见变异(MAF>5%)和 60% 的稀有变异(MAF> 2.5%),适合全基因组关联研究(GWAS)。
▲采用 Illumina 专利的 BeadArrayTM 技术,可提供非常高的数据质量,平均检出率 >99%,重复率 >99.9%。
规格:8 样本 / 芯片。
位点数:SNP 位点超过 90 万。
3、Axiom®Precision Medicine Research Array(PMRA)——精准医疗研究芯片
作为一款高密度 SNP 芯片,SNP 标记平均覆盖了全基因组,可保证 PMRA 满足全基因组关联分析和遗传连锁分析的需要。另外,PMRA 囊括了新的 GWAScatalog 数据库中的 SNP 位点,体现了几乎最新的临床研究成果,可担任起易感位点检测和基因分型的任务,提供用药和治疗方案上的指导。
▲PMRA 提供来自新的 1000Genome PhaseIII 和更新至 2016 年 5 月份的 NHGRI-EBIGWAS catalog 数据库中的内容,为关联分析提供更精准的标记;
▲囊括已经临床验证的致病位点;
▲覆盖来自 GWAS 研究的癌症相关常见变异(commonvariants);
▲覆盖了免疫和转移相关的变异,包括 HLA 和 KIR 相关的 marker,并可使用 AxiomHLA 分型软件进行 HLA 分型;
▲包括了血细胞表型相关的 marker,和血液病学 GWAS 研究中得到的结果;
▲与其它分型平台共享了一些 fingerprintSNPs,可用于追踪样本。
规格:96 样本 / 芯片。
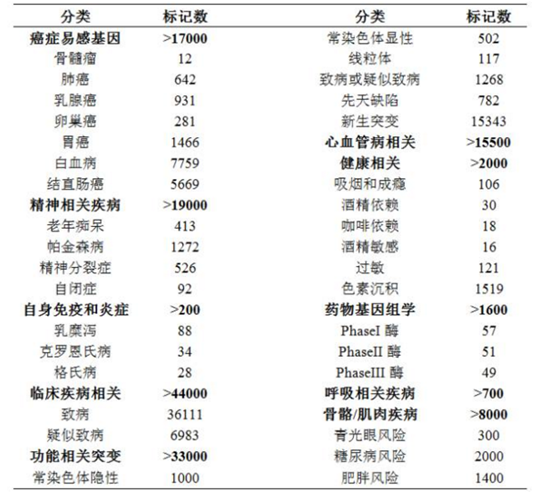
PMRA 芯片 SNP 疾病易感位点标记数:
4、Illumina GSA 芯片(Infinium Global Screening Array)
Illumina 公司的 GSA (Global Screening Array) 芯片是一款先进的基因分型芯片,包含 66 万个探针,与疾病、注释、性状有关的位点有 132,262 个,为群体规模的遗传研究、变异筛查和精准医学研究提供了一种经济的解决方案。
▲全面的位点信息
Infinium Global Screening Array-24 v2.0 BeadChip 结合了多种族全基因组内容,精选临床研究变体和质量控制(QC)标记,用于精准医学研究。
所有 26 个 1000G 群体中的 MAF> 1% 的位点。基于 ClinVar,NHGRI,PharmGKB 和 ExAC 数据库的临床研究内容,包括具有已建立的疾病关联的变异,相关的药物基因组学标记。
▲广泛的适用性
在全球范围内采用该芯片的样本超过 550 万个,可以提供人类疾病研究人员,医疗保健网络,消费者基因组学公司和基因组服务提供商网络,以帮助发现基因信息。
▲广泛的临床研究应用
该阵列为临床研究应用提供基因组工具,包括疾病风险分析研究,药物基因组学研究,健康评估和复杂的疾病。2018 年 3 月,Illumina 发布了免费更新,更新覆盖了药物基因组学,ClinVar,HLA 和 ACMG 中的更多位点。
▲稳健的分析和高吞吐量的工作流程
该阵列支持基因分型和 CNV 呼叫样本类型,包括唾液,血液和口腔拭子。该格式还可以每周处理数千个样本,并进行为期 3 天的人口规模研究工作流程。
5、Illumina Asian Screening Array(ASA)
这款芯片在继承了 GSA 芯片超高性价比的基础上,在位点上采用了亚洲人群的数据,并对 CNV 探针进行了升级,可谓是 GSA 芯片的亚洲定制 PLUS 版。
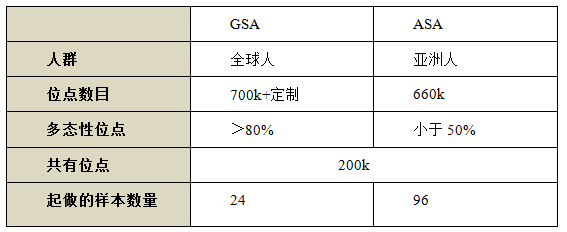
ASA 位点信息
70 万个标记,覆盖东亚及东南亚人群参考基因组;
基因组骨架基于 9000+ 亚洲人全基因测序数据(15-30X)优化获得;
更适合用于亚洲人基因组 imputation;
约 15 万个临床研究、疾病探索、质控等标记;
经济高效,多态性位点达 80% 以上 ;
广覆盖遗传性疾病相关基因;
迄今覆盖亚洲人稀有变异(MAF1-5%)的芯片。
特色位点
高覆盖已知可解释遗传变异——Clinvar 数据库 。
CNV 探针升级:可进行 100 多种疾病,2,258 个基因 4000 多个 CNV 结构变异检测。
遗传病基因广泛覆盖:涵盖美国医学遗传学与基因组学学会(ACMG)推荐的 58 个基因,用于遗传病研究。
药物基因组研究芯片:涵盖 CYP2D6、G6PD、CYP2C9、SLCO1B1、CYP 2C19 等常见药物代谢基因的 373 个 star alleles 和 CNV。
芯片优势
应用广:几乎涵盖目前可解释的所有 SNP 位点。
通量高:70 万 SNP/ 样本,每周 2000+ 样本。
性价比高:多态性位点达 80% 以上,价格低。
灵活性强:可实现不同研究方向特色定制。
芯片应用
研究亚洲人心脑血管、肿瘤、神经系统疾病等复杂疾病易感位点及 biomarkers。
解析遗传病、新生儿缺陷等疾病的发生机制。
肿瘤、心血管、代谢类疾病等染色体结构变异研究。
药物基因组学研究。
追溯祖源。
样本要求
血液样本:EDTA 抗凝,2ml,-20℃冰箱保存;
新鲜冰冻组织:液氮冷却、-80℃冰箱保存;
样品量要求:DNA 样品浓度:≥50ng/µl;OD260/280=1.8-2.0;DNA 样品总量:≥1ug。
应用案例
基于候选基因策略,利用 SNP 单倍型数据库,识别 Tag SNP,应用 GoldenGate 定制芯片开展冠心病的研究。伯豪生物挑选 2000 多个与可能导致冠心病的候选基因相关的 SNP,对患者和对照人群共近 3000 份样本进行了基因分型,对数据进行的关联分析发现了一些与疾病有关的易感位点。2009 年伯豪生物与上海高血压研究所合作开展高血压的全基因组关联研究。
与美国 Rutgers 大学合作,利用 Illumina 的技术平台,采取候选基因策略,利用 GoldenGate 定制芯片开展了自闭症关联分析研究,探讨这类疾病的发病机理及遗传易感性。
与吸烟导致肺癌相关的遗传因素、基因 - 环境互作机制:吸烟人群和长期在粉尘环境中工作的人群,肺癌的发病率高,伯豪生物以此为模型,收集患者的 DNA 样本,应用 GoldenGate 定制芯片进行关联分析,研究遗传多态与吸烟易患肺癌风险的相关性,筛选到一系列吸烟人群易发肺癌的易感基因位点,鉴定出在特定环境暴露下决定患病风险的易感基因,从分子水平研究环境化学物质与基因的互作,从而将病原学中基因 - 环境互作机制用于流行病学研究,确定高危人群。
早在 2008 年安徽医科大学,利用伯豪生物的研发公共服务平台的综合技术力量,就银屑病、白癜风和系统性红斑狼疮在中国人群的遗传易感性进行研究,先后发表了几篇 Nat Genetic; 2009-2010 年,与山东皮肤病性病防治研究所合作开展了麻风病的全基因组关联研究和家系连锁分析,取得的轰动性发现发表在国际顶级医学杂志《新英格兰医学杂志》;2010 年伯豪生物与上海内分泌研究所合作,对 GD 和糖尿病进行全基因组关联研究。

