
Hieff NGS® ATAC-Seq Library Prep Kit for Illumina® ATAC建库试剂盒
产品名称: Hieff NGS® ATAC-Seq Library Prep Kit for Illumina® ATAC建库试剂盒
英文名称: Hieff NGS® ATAC-Seq Library Prep Kit for Illumina® ATAC建库试剂盒
产品编号: 12208ES
产品价格: 询价
产品产地: Yeasen
品牌商标: Yeasen
更新时间: 2025-11-07T19:46:48
使用范围: null
- 联系人 : 李自转
- 地址 : 上海市浦东新区天雄路166弄一号楼三层南单元
- 邮编 : 200030
- 所在区域 : 上海
- 电话 : 139****5640 点击查看
- 传真 : 点击查看
- 邮箱 : lizizhuan@yeasen.com
- 二维码 : 点击查看
样本即用型试剂盒:含ATAC-seq实验中除样本以外的所有试剂,无需额外其他组分。
组织样本需搭配12514/12516ES(组织细胞核提取试剂盒)。
植物样本需自行处理成细胞核样本后再搭配12208使用。
Hieff NGS® ATAC-Seq Library Prep Kit for Illumina®是针对Illumina®高通量测序平台研发的用于ATAC-Seq实验的文库构建试剂盒,适用于100-100,000个细胞起始量的样本建库。ATAC-Seq(Assay for Transposase Accessible Chromatin with high-throughput sequencing)技术能够利用Tn5 转座酶识别染色质开放区域,快速有效的反映表观遗传状态。经过细胞收集及裂解、转座酶片段化、磁珠回收片段化DNA、文库扩增和磁珠分选等步骤,DNA片段最终转化为适用于Illumina®平台测序的文库。
本试剂盒包含两个独立模块:BOX-I和BOX-II。BOX-I为提取DNA的DNA Extract Beads和回收文库的DNA Clean Beads,BOX-II包含细胞裂解、片段化以及后续文库扩增所需的所有试剂。此外,本试剂盒已在不同种类样本(如K562、MCF-7细胞,小鼠肝脏等)中进行了验证,均具有良好的建库效率和建库产量。本试剂盒提供的所有试剂都经过严格的质量控制和功能验证,最大程度上保证了文库的稳定性和重复性。
●含两种纯化磁珠:针对小片段纯化,回收效果更好。
●实验操作简单、实验周期短:建库一步法,节省建库时间;
●样本投入量低,投入量兼容范围广:能兼容100~10万个细胞投入。
●实验重复性好
产品应用:ATAC-seq、染色质开放性研究、转录因子鉴定、足迹分析等
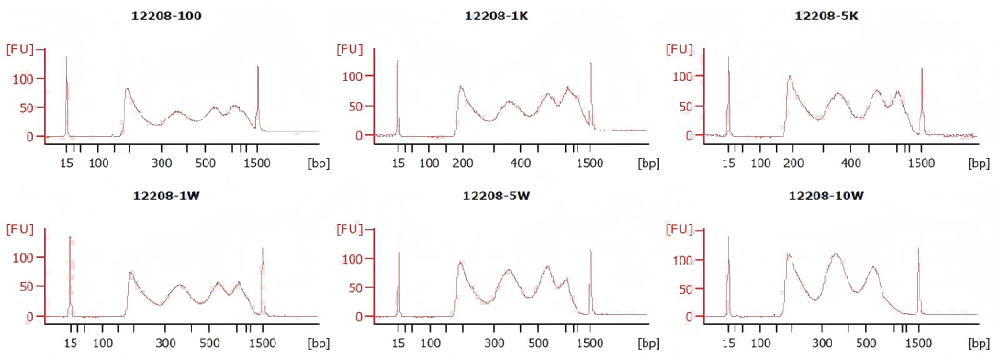
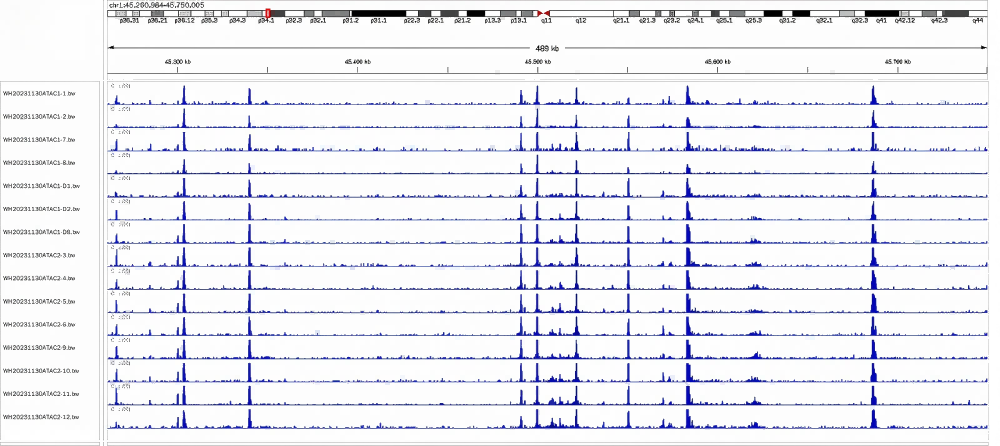
1、不同细胞投入量的ATAC建库结果一致性较高
293细胞ATAC建库,细胞投入量从100个到10万个,不同细胞投入量建库后,文库峰型以及IGV可视化结果一致性较高。
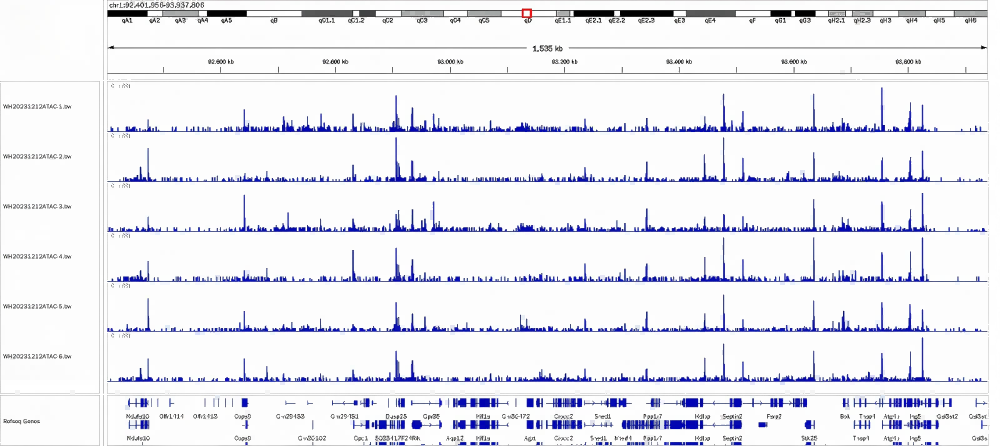
2、冻存样本ATAC建库结果展示
对不同冻存时间的样本进行ATAC建库,目前测试冻存20天内的样本建库无问题,且测序结果信号明显。
冻存样本要求:尽量客户样本保存在液氮中,如果保存-80℃冰箱,需要无反复冻融情况。

BOX-I:2~8℃保存。
BOX-II:-25~-15℃保存。
Q:投入多少细胞量合适?
A:兼容100-100000个细胞或细胞核,最佳投入量为5w个细胞或细胞核。
Q:ATAC文库产量低的原因?
A:1. 细胞核投入量少或实验过程中细胞核部分丢失。细胞核离心后弃去上清液时容易造成丢失,在离心时,统一EP管方向并标记细胞沉积位置,弃上清时避免碰到沉淀,在实验操作过程中避免剧烈涡旋振荡;2. 转座酶切割不充分,这种原因有很多,首选需要确认细胞核的纯度,如果杂质较多,可能影响转座酶的效果,提取细胞核时尽量多清洗几遍,以获得纯净的细胞核。
Q:TSS富集程度不好是什么原因?如何增加?
A:TSS富集效果不好通常是由于实验样本细胞活性不好或游离DNA较多。可对细胞或细胞核进行镜检确认细胞或细胞核完整性。
2、ATAC文库中的线粒体reads较多,如何减少?
A:通常是由于细胞核提取过程中,细胞漂洗时未能吸出所有的上清液(其中包含mtDNA)。应吸尽上清,同时注意不要影响到细胞核完整性。或者可以多清洗几遍,提升细胞核的纯度。
